本文将分享CSC1001:IntroductiontoComputerScience的详细内容,此外,我们还将为大家带来关于AIforScience交流会来了!科学计算前沿邀您共同探讨、AI+Scie
本文将分享CSC1001: Introduction to Computer Science的详细内容,此外,我们还将为大家带来关于AI for Science 交流会来了!科学计算前沿邀您共同探讨、AI+Science 系列(二):国内首个基于 AI 框架的 CFD 工具组件!赛桨 v1.0 Beta API 介绍以及典型案例分享!、AI+Science 黑客马拉松|赛程发布|10万奖金等你来拿!、AI+Science:基于飞桨的 AlphaFold2,带你入门蛋白质结构预测的相关知识,希望对你有所帮助。
本文目录一览:- CSC1001: Introduction to Computer Science
- AI for Science 交流会来了!科学计算前沿邀您共同探讨
- AI+Science 系列(二):国内首个基于 AI 框架的 CFD 工具组件!赛桨 v1.0 Beta API 介绍以及典型案例分享!
- AI+Science 黑客马拉松|赛程发布|10万奖金等你来拿!
- AI+Science:基于飞桨的 AlphaFold2,带你入门蛋白质结构预测

CSC1001: Introduction to Computer Science
CSC1001: Introduction to Computer Science
Programming Methodology
Assignment 3
Assignment description:
This assignment will be worth 9% of the final grade.
You should write your code for each question in a .py file (please name it using the
question name, e.g. q1.py). Please pack all your .py files into a single .zip file, name it using
your student ID (e.g. if your student ID is 123456, then the file should be named as
123456.zip), and then submit the .zip file via Moodle.
Please also write a text file, which provide the details about how to run your code for each
question. The text file should be included in the .zip file as well.
Please note that, the teaching assistant may ask you to explain the meaning of your
program, to ensure that the codes are indeed written by yourself. Please also note that
we may check whether your program is too similar to your fellow students’ code using
Blackboard.
This assignment is due on 5:00PM, 14 Apr (Sunday). For each day of late submission, you
will lose 10% of your mark in this assignment. If you submit more than three days later
than the deadline, you will receive zero in this assignment.
Question 1 (20% of this assignment):
Write a Python class, Flower, that has three instance variables of type str, int, and float,
that respectively represent the name of the flower, its number of petals, and its price.
Your class must include an initializer that initializes each variable to an appropriate value,
and your class should include methods for setting the value of each type, and retrieving
the value of each type. Your program should be robust enough to handle possible
inappropriate inputs.
CSC1001 作业代做、代写 Programming Methodology 作业、代做 Python 实验作业、Python 程序设计作业调试
Question 2 (40% of this assignment):
Write a Python class that inputs a polynomial in standard algebraic notation and outputs
the first derivative of that polynomial. Both the inputted polynomial and its derivative
should be represented as strings.
For example, when the inputted polynomial is , the output of
your program should be .
Note: (1) The inputted polynomial will contain only one variable, and the variable is not
necessarily ‘x’; (2) In the inputted polynomial, the terms are not necessarily arranged in
descending or ascending orders.
Question 3 (40% of this assignment):
Write a Python class to simulate an ecosystem containing two types of creatures, bears
and fish. The ecosystem consists of a river, which is modeled as a relatively large list.
Each element of the list should be a Bear object, a Fish object, or None. In each time
step, based on a random process, each animal either attempts to move into an adjacent
list location or stay where it is. If two animals of the same type are about to collide in
the same cell, then they stay where they are, but they create a new instance of that
type of animal, which is placed in a random empty (i.e., previously None) location in the
list. If a bear and a fish collide, however, then the fish dies (i.e., it disappears).
Write an initializer for the ecosystem class, the initializer should allow the user to assign
the initial values of the river length, the number of fishes and the number of bears.
Before the simulation, fishes and bears should be allocated randomly into the river. The
ecosystem class should also contain a simulation() method, which will simulate the next
N steps of the random moving process. N should be inputted by the user. In each step of
your simulation, all animals in the river should try to take some random moves. In each
step of your simulation, the animals should take actions one by one. The animals on the
left will take actions first.
For example, assume that before the simulation, the initial state of the river is:
In which, ‘F’, ‘B’ and ‘N’ denote fish, bear and empty location respectively. Assume that
in the first step of simulation, the first fish will move to the left, the first bear will move
to the right, and the second bear will remain still. Then after the first step, the state of
the river is:
To generate random numbers in Python, you should import the random() function by
using the following statement:
By assigning the return of the random() function to a variable, you will get a random
floating point number in the range of [0, 1]. The following code is an example of using the
random() function:
因为专业,所以值得信赖。如有需要,请加 QQ:99515681 或邮箱:99515681@qq.com
微信:codinghelp

AI for Science 交流会来了!科学计算前沿邀您共同探讨
随着深度学习不断驱动技术创新,人工智能科学计算迈向高质量发展道路。百度飞桨作为科学计算的坚定支持者,计划于 7 月 13 日举办飞桨科学计算线下交流会。本次交流会以百度飞桨深度学习框架为基座,广泛联动人工智能科学计算领域头部专家学者、高等院校、科研机构和开发者,搭建科学计算从业者广泛交流、深度融合的平台,讨论行业动态、科研成果、开源建设等多种议题,全力推进 AI for Science 建设与发展。
在这次交流会上,我们将向大家介绍飞桨 PaddleScience v1.0 版本。赛桨 PaddleScience 是一个应用于科研领域的端到端科学计算工具组件,利用飞桨深度学习框架的自动 (高阶) 微分机制和高层 API,解决物理、化学、气象等领域的问题。支持物理机理驱动、数据驱动、数理融合三种求解方式,并提供了基础 API 和详尽文档供用户使用与二次开发。
活动详情
活动主题
AI for Science— 人工智能加速科学发现与科学计算
活动时间
7 月 13 日 14:00-17:00
活动地点
上海・百度飞桨赋能中心
报名要求
欢迎从事计算机 / 人工智能或各领域科学计算方向的研究人员踊跃报名
报告主题
本次线下交流会我们邀请到国内外优秀的科研人员分享他们在相关领域的知识和研究成果,为大家提供一个学习和深入探讨的机会。
主题一:飞桨科学计算产品报告
主讲人:张艳博,飞桨高级技术产品经理
演讲主题:《飞桨 AI for Science 领域探索与产品建设现状》
报告简介:介绍飞桨 AI for Science 整体产品建设情况
主讲人:何森森,飞桨工程师
演讲主题:《飞桨
报告简介:介绍飞桨 AI for Science 整体产品建设情况
主题二:AI for Science 学术报告
主讲人:孟旭博,2017 年博士毕业于华中科技大学能源与动力工程学院;2018 年 - 2022 年美国布朗大学应用数学系从事博士后研究工作,合作导师为美国工程院院士 George Em Karniadakis 教授;2022 年 3 月至今任华中科技大学数学与统计学院数学与应用学科交叉创新研究院副教授。主要研究方向为科学计算中的深度学习方法。截至目前已在 JCP、CMAME、SIAM Review 等期刊发表 SCI 论文 20 余篇,谷歌学术总引用 2600 余次,4 篇论文入选 ESI 高被引论文,1 篇论文为热点论文;担任 JCP、SISC、CMAME、Nat. Comput. Sci . 等期刊审稿人。
演讲主题:《科学计算:融合多保真数据的复合神经网络》
报告简介:机器学习的最新发展也影响了物理系统的计算建模,例如地球科学和工程学。通常,复杂物理系统的优化需要大量高保真数据集,这可能导致计算成本过高。另一方面,不充分的高保真数据会导致不准确的近似值和可能的错误设计。多保真度建模已被证明可以通过利用低保真度和高保真度数据在不同应用中实现高精度。在本次演讲中,我将介绍一种新开发的用于多保真度数据融合的深度学习算法及其在函数逼近和逆 PDE 问题中的应用。
主讲人:王韫博,上海交通大学助理教授,清华大学博士,CCF 优博,国家自然科学基金原创探索项目负责人。主要从事机器学习与计算机视觉的研究,专注于可微物理模拟、有模型强化学习等方向,在 TPAMI、NeurIPS、ICML、CVPR 等 CCF-A 类期刊和会议上发表论文 20 余篇,2020 年入选上海市青年科技扬帆计划。
演讲主题:《世界模型:直觉物理推理与决策》
报告简介:世界模型,是指智能体通过与环境交互,或通过观察物理世界的视觉表观演变,探索和推断其内部动力学模型,构建物理世界的可微分模拟器。近十年间,J. Tenenbaum、J. Schmidhuber、Y. LeCun 等人分别从贝叶斯认知理论、视觉环境决策、自监督学习的角度给出了世界模型的不同表达形式。然而,如何在复杂的视觉场景中建立有效的世界模型仍然是一项开放课题。本次报告围绕世界模型的 “观察” 与 “交互” 两个方面,分别介绍 “3D 逆图形学物理推断” 和 “基于世界模型的强化学习” 方法,相关工作分别发表于 2022 年的 ICML 和 NeurIPS(Spotlight)。前者为流体动力学模拟提供了一种新思路,后者应用于 DeepMind Control 机械臂视觉控制任务和 CARLA 自动驾驶任务,为当前最佳基线模型之一。
主讲人:Madeleine Martinsen, Head of R&D Hoist & Underground Mining at ABB Service. More than 34 years of wide competences from engineering, internal auditing, controlling to management positions within both the Power and Automation Industry at ABB. Including abroad assignments in Germany, Ecuador, USA and Denmark. A trouble-shooter with successful experience from “turning around” businesses. As a person enthusiastic, she is open minded, optimistic, curios, analytic, structured and result oriented. Her driving force is best used within an organisation where daily operational excellence is at focus as for serving customers. Currently focusing on finalizing her PhD with the title ‘Monitoring of airflow and airborne particles, to provide early warning of irrespirable atmospherics conditions’.
主讲人:Erik Dahlquist (瑞典皇家工程院院士), Experienced Professor with a demonstrated history of working in the higher education industry. Skilled in Computer Science, Smart Grid, Biomass, Modeling, and Research and Development (R&D). Strong education professional with a PhD focused in Chemical Engineering from Kungliga tekniska högskolan.
演讲主题:《工业数字化:Smart Industries demands Smart Services》
报告简介:The industries production is predicted to completely develop into an autonomous operation in the future. Developing an autonomous manufacturing system can for some industries prove difficult due to inadequate connectivity and non-uniform manufacturing environment. This is the case for the mining business. To resolve the connectivity issue, the industries are investing in information system (IS) and information technology (IT), infrastructure to provide connectivity to the overall control system, which will support their digitalization journey. Anyhow, the transition to a fully autonomous mining will not happen over a night. A complete autonomous mining operation will demand solutions that can be trustworthy. Timely detection of faults, which can lead to harmful and dangerous situations, is of the most importance in an autonomous mining operation. Preventing and predicting the need for maintenance will play an important ingredient succeeding.
日程安排

AI+Science 系列(二):国内首个基于 AI 框架的 CFD 工具组件!赛桨 v1.0 Beta API 介绍以及典型案例分享!

AI for Science 被广泛认为是下一代科研范式,可以有效处理多维度、多模态、多场景下的模拟和真实数据,解决复杂推演计算问题,加速新科学问题发现 [1] 。百度飞桨科学计算工具组件赛桨 PaddleScience 是国内首个公开且可应用于 CFD(Computational Fluid Dynamics,计算流体力学)领域的工具,提供端到端应用 API,致力于解决科学计算类任务。赛桨综合数学计算与物理数据相结合的处理方法,提供物理机理约束的 PINNs(Physics Informed Neural Networks 物理信息神经网络)加速求解偏微分方程,解决计算流体力学中的仿真分析。本篇文章将重点介绍赛桨 PaddleScience v1.0 Beta 的典型案例及 API 使用示例。
赛桨提供的典型案例包含使用 AI 方法进行顶盖驱动方腔流(LDC)、达西流、2D&3D 圆柱绕流的流场预测及涡激振动(VIV)。所有案例都基于泛化的微分方程、PINNs 求解器、控制体、网络定义以及可视化等多种接口。下面我们将详细讲解赛桨 PaddleScience v1.0 Beta 中提供的计算流体力学案例及科学计算 API 的功能及使用。
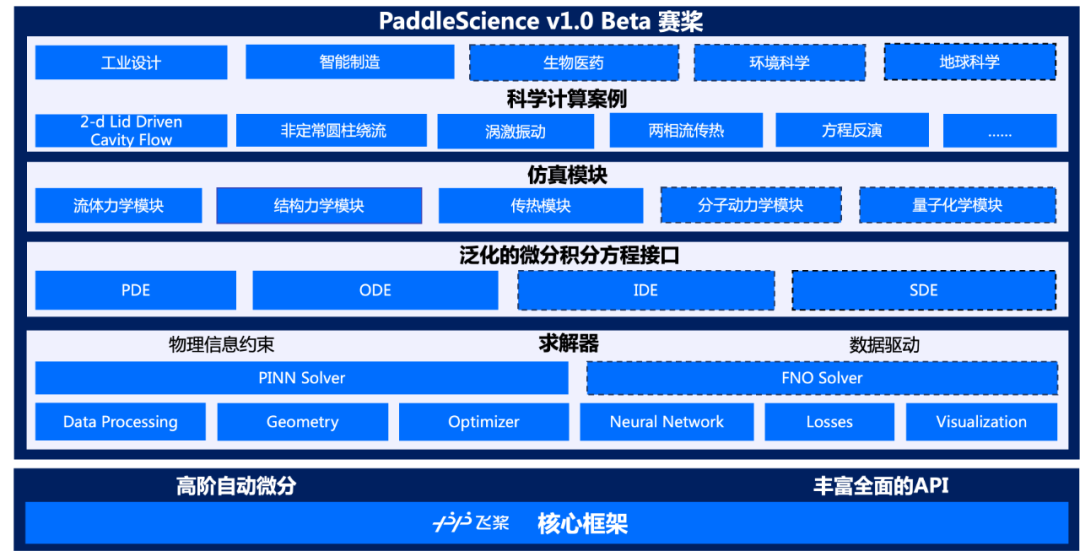
图 1 PaddleScience v1.0 Beta 产品全景
场景案例详解
前言
在流体领域,流体力学作为比较传统的物理学科,广泛应用于航空航天、船舶工业以及建筑、水利、能源等工程领域。
-
在航空工程和造船工业中,如飞行器和船舶的外形设计、操控性、稳定性等问题成为流体力学中广泛的研究课题,并促使流体力学得到了很大的发展。
-
在水利工程中,如大型水利枢纽和水力发电站的设计与建造、洪峰的预报工作、河流泥沙等问题都与流体力学紧密关联。
-
在动力机械制造工业中,如何提高水力及蒸汽涡轮、喷气发动机、压缩机和水泵等动力机械的性能,与叶片、导流片及其他零件设计形状的正确性有关。
随着工程问题的深入,流体力学已经逐渐与其他学科互相交叉渗透,形成新的交叉学科或边缘科学,如磁流体力学、物理 - 化学流体力学、生物流变学等等。
围绕不同的流体问题,当前流体力学分析主要基于数值计算。典型的方法有直接数值模拟 (DNS) 、雷诺平均方法 (RANS)、大涡模拟方法 (LES)、分离涡模拟 (DES) 以及格子玻尔兹曼法(LBM)等求解流体本构 N-S (Navier-Stokes) 方程。由于物理问题的复杂度,数值方法往往有很多局限性,如计算效率等。PaddleScience 的 PINNs 科学计算求解器,将物理信息融入神经网络,针对流体力学问题提供新的解决思路。本章节中,我们将介绍 2D&3D 圆柱绕流及涡激振动的案例,展示 PaddleScience 的基础科研能力。

图 2 不可压缩 N-S (Navier-Stokes) 方程
2D 非定常圆柱绕流
圆柱绕流作为经典的 CFD 问题,在不同的雷诺数下,涡脱落产生的卡门涡街类型不同,是能够综合体现层流、湍流过渡的典型问题。针对该问题,采用 PINNs 方法,并基于连续时间的 2D 不可压、非定常 NS 方程作为约束深度学习神经网络的物理规则,将传统的 CFD 求解转换为神经网络参数的优化问题。同时,为了加速训练的收敛时间,提高预测精度,采用半监督方式,从开源 CFD 工具 OpenFOAM 的结果中记录边界位置处约 200 个测点在不同时刻的流场信息,与 N-S 方程、初边值条件等共同形成了网络优化的损失函数。对雷诺数 Re=100 工况,定义约 110W 个时空训练点(t, x, y),并采用 NVIDIA V100-32G 单卡训练约 8 小时,结果如下图所示。基于 PINNs 方法构建的网络能够完整的模拟卡门涡的周期性脱落,且预测的流场结果与 OpenFOAM 相对误差小于 5%(除边界层中个别点外),可满足工程需求。
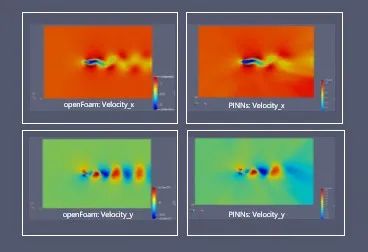
图 3 Re=100 的 2D 圆柱绕流结果
(左:OpenFOAM 结果,右:PINNs 方法结果)
同时,基于训练完成的模型,利用推理功能,8ms 内即可完成百万级空间位置在 30 个时间步的结果预测,相比于同样网格数量且固定求解配置的 OpenFOAM 计算过程,速度提升近 12000 倍,非常利于后期在线评估、优化等工程应用。
- 案例链接:
https://github.com/PaddlePaddle/PaddleScience/tree/develop/examples/cylinder/2d_unsteady_continuous
3D 非定常圆柱绕流
除 2D 圆柱绕流外,赛桨 PaddleScience 同时提供 3D 圆柱绕流案例,基于 PINNs 方法,求解 3D 非定常不可压缩 Navier-Stokes 方程,完成了无模化 Re=100 的圆柱绕流问题求解。在该问题中,采用 OpenFOAM 的结果作为基准,选择流场稳定的相对 0 时刻,并记录有限时间内特定测点的数据。采用离散时间的 PINNs 神经网络进行训练,其模型数量与所选取的时间步长相关,在 t_start 至 t_end 内,定义离散时间步长 dt,采用 (t_end-t_start)/(T*dt) 个模型进行训练,其中 T 为涡脱周期。对比 OpenFOAM 的理论值,相对误差在 5% 以内。x 轴上的流场速度变化如下:

图 4 3D 圆柱绕流 x 轴流场速度
- 案例链接:
https://github.com/PaddlePaddle/PaddleScience/tree/develop/examples/cylinder/3d_unsteady_discrete
涡激振动
涡激振动(VIV)是一种典型的流致振动,是流体经过结构后稳定涡脱频率与结构固有频率综合的流固耦合问题。是海洋工程中立管、输油管路等大跨度结构的主要损伤源,但由于结构复杂,无法有效测量刚度、阻尼等本构特征,导致损伤探测及预防难度较大。相比基于 PINNs 方法实现 2D 圆柱绕流正问题分析,解决 “反问题” 是 PINNs 方法的一个亮点,可通过部分实验数据 “逆向” 预测流场中结构的刚度、阻尼等本构特征,从而对实际工程中的复杂结构的疲劳损伤及破坏的预测及预防提出指导。
从工程落地的角度,赛桨 PaddleScience 从 “半实物仿真” 的技术路线出发,构建了涡激振动试验台架以及模型驱动试验装备的电控闭环,创新性地探索并论证深度学习模型与物理装备的虚实结合的技术可行性。
通过对涡激振动开展分阶段分析,首先基于加速度计及激光设备等传感器采集结构振动振幅与结构升力,对系统进行弹簧 - 振子单自由度等效。基于实测的 160 组位移及受力数据,训练过程中最小化 “振幅”、“升力”、“方程” 等共同组成的损失函数。采用 V100-16G 单卡训练约 0.5h,预测结果如下图(右上)。基于 PINNs 预测的结构振动振幅、结构升力结果与试验数据的相对误差均在 2% 以内。同时基于 “反问题” 方式分析得到的结构刚度、阻尼分别为 1.092964 与 4.1e-6,相比真实值 1.09 与 0,相对误差均小于 2%。
涡激振动主要基于试验与深度学习模型结合的方式进行,通过构建风洞试验装备,在第一阶段完成结构刚度、阻尼的预测,在第二阶段则基于得到的结构刚度、阻尼等属性,进行流场重构以及升阻力的预测。过程中基于赛桨提供的泛化 PDE 接口,对涡激振动中流固耦合方程也重新进行了整合,定义新的网络与求解过程,具体流程如下图所示。
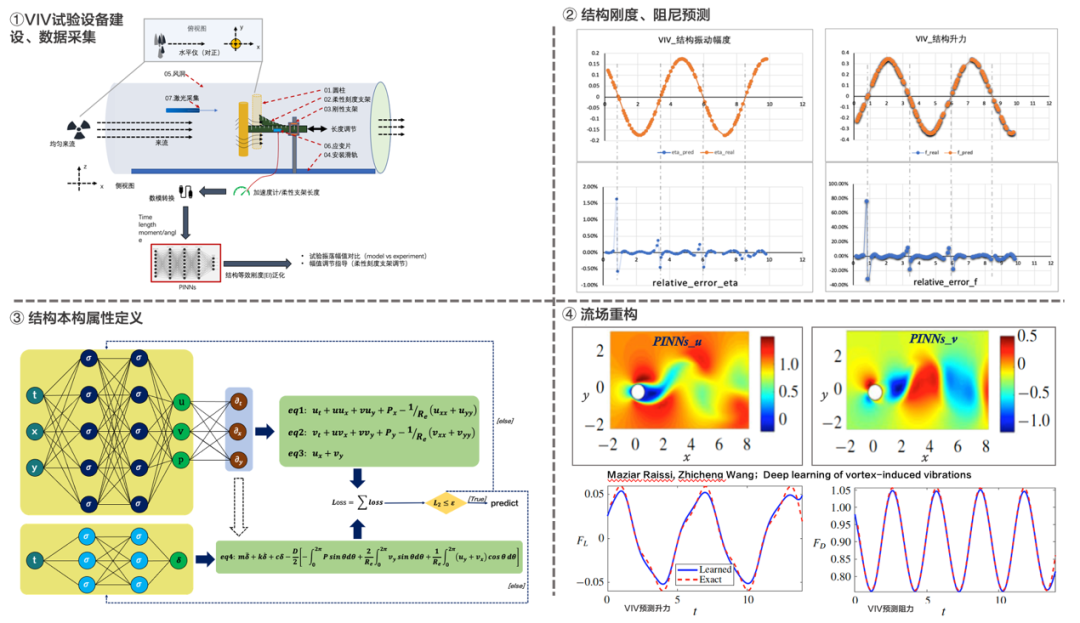
图 5 VIV 试验及深度学习模型联合验
- 案例链接:
https://github.com/PaddlePaddle/PaddleScience/tree/develop/examples/fsi
API 功能及使用示例
上述案例充分利用了赛桨 PaddleScience v1.0 Beta 提供的 API。本部分将着重介绍涉及的主要 API 接口及使用示例方法。
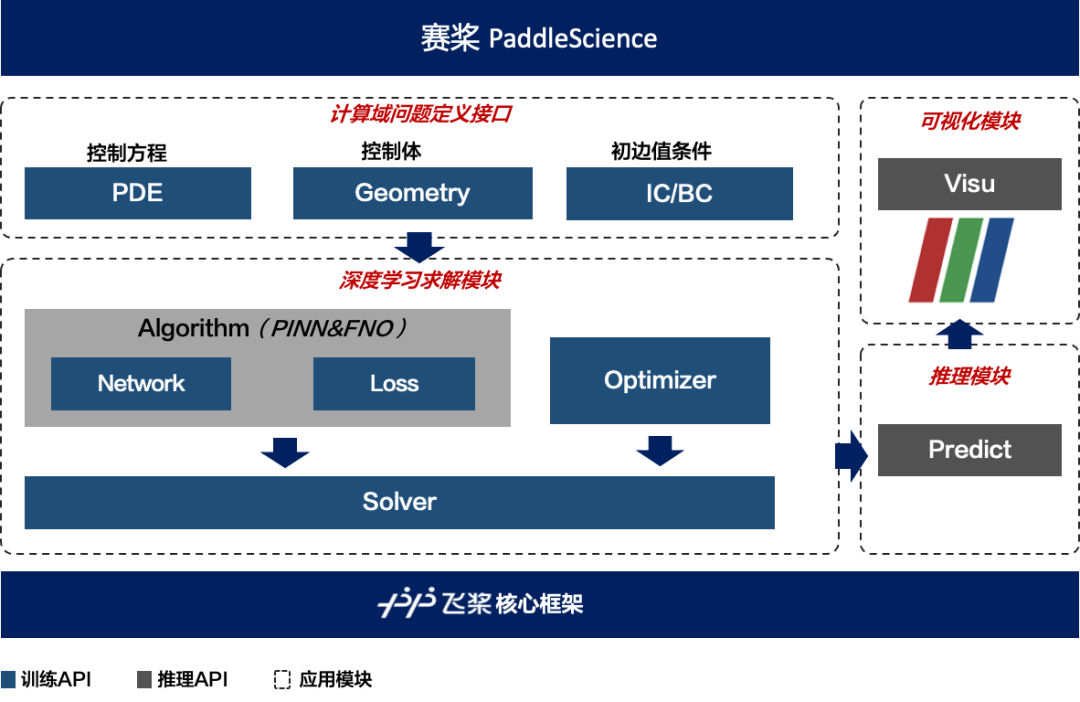
图 6 PaddleScience 科学计算工具组件设计架构
科学计算问题定义
科学计算问题定义包含三部分:方程定义(PDE)、计算域定义(Geometry)、初边值条件定义(IC/BC)。下图展示了如何定义在一个方形区域内求解二维非定常 Navier-Stokes 方程问题。具体而言,该问题数学上由 (图 b) 定义,包含方程、初边值条件及计算域信息,代码上每一条信息由相应接口描述(图 a 及图 c)。图中展示了一部分数学信息和接口的对应关系,该对应关系由同样的颜色方框表示。

图 7 PaddleScience 科学计算问题定义
本章节将介绍关于科学计算问题定义相关的 API。
偏微分方程(PDE)
赛桨支持调用预置方程接口及自定义方程接口。
-
预置方程接口:方程定义内置在赛桨中,用户直接调用即可,仅需配置相关参数(如维度、物理属性等)。
-
自定义方程接口:用户使用 Python SymPy 定义自变量、因变量及方程,通过 “add_equation” 接口将方程加入 PDE 模块。


对于高维偏微分方程,连续时间方法将时间和空间均视为网络的输入;离散时间方法首先使用数值 (隐式) 方法对时间离散,得到仅包含空间变量的方程,进而使用 PINNs 方法求解该方程,3D N-S 方程及使用隐式离散方法得到的方程如下,其中 n 时刻的状态(3 个方向的速度)已知,求解 n+1 时刻的状态(3 个方向的速度)。
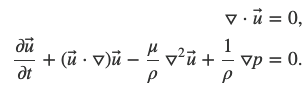
图 8 使用隐式方法对时间离散后得到的方程
如下代码展示如何定义一个非定常 N-S 方程,以及如何使用连续时间方法和离散时间方法,进行方程离散。仅需通过改变参数 “time_method” 的取值即可选取对应的方法。

计算域(Geometry)
赛桨提供了常用的计算域形状,如立方体、立方体除去圆柱等几何外形,及 VTK 可视化支持,同时提供 "add_boundary" 接口定义边界。

初边值条件(IC/BC)
边值条件模块预置了 Dirichlet/Neumann/Robin 边值条件,仅需定义边值条件,并通过 “set_bc” 接口将其指定到对应边界即可。对于 PDE,以类似方式增加了初值条件模块。

深度学习方法求解模块
上一章节介绍了如何使用赛桨定义科学计算问题,本章节继续介绍深度学习方法求解上述科学计算问题所需使用到的模块。
网络(Network)
赛桨支持全连接网络(FC)以及初始化网络权重的接口 initialize,支持从文件读取权重进行初始化,以及使用飞桨预置的初始化函数(paddle.nn.initializer)进行初始化。

损失函数(Loss)
赛桨提供了灵活的 Loss 设置方式,允许通过设置独立的权重系数配置多任务 Loss (Loss = w_1 * Loss_equation + w_2 * Loss_ic+ w_3 * loss_bc +w_4 * Loss_data)。

优化器(Optimizer)
赛桨支持 Adam 优化器。

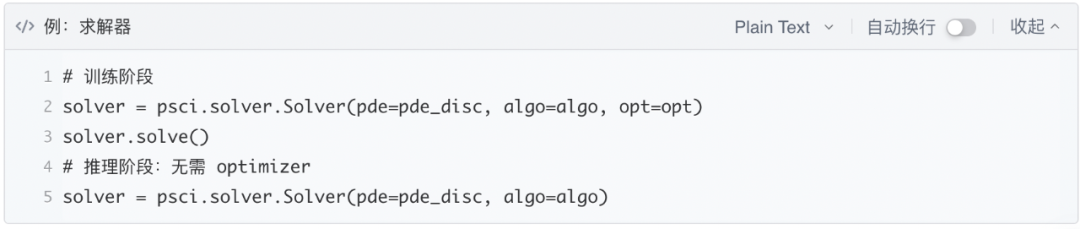
求解器(Solver)及求解模式
求解器提供了控制功能,在训练和推理阶段可以分别使用。
启动自动并行模式
使用自动并行技术实现分布式计算内嵌在赛桨中,无需特别设置,使用如下代码运行程序即可实现数据自动并行。

启动动态图 / 静态图 / 自动微分模式
赛桨后端支持动态图和静态图模式,可以通过简单的接口进行切换。另外,在单机静态图模式下,赛桨提供接口启用高阶自动微分。

下一期我们将对支持 PaddleScience 的底层框架技术,如自动微分机制、编译器等功能进行详细介绍,敬请期待~
- 引用:
[1]《IDC perspective:AI for Science 市场研究》报告正式启动.
https://mp.weixin.qq.com/s/gtEbuSULI5fzCIvbDKhkfA
- 拓展阅读:
1.《AI+Science 系列(一):飞桨加速 CFD(计算流体力学)原理与实践》
2. 赛桨 PaddleScience v1.0 Beta:基于飞桨核心框架的科学计算通用求解器
- 相关地址:
1. 飞桨 AI for Science 共创计划:
https://www.paddlepaddle.org.cn/science
2. 飞桨 PPISG-Science 小组:
https://www.paddlepaddle.org.cn/specialgroupdetail?id=9
关注【飞桨
获取更多技术内容~
本文同步分享在 博客 “飞桨 PaddlePaddle”(CSDN)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与 “OSC 源创计划”,欢迎正在阅读的你也加入,一起分享。

AI+Science 黑客马拉松|赛程发布|10万奖金等你来拿!
点击左上方蓝字关注我们

“机器学习与物理建模的结合正在改变着科学研究的范式。那些希望通过计算建模突破科学边界、解决困难问题的人们正在以前所未有的新方式集结起来。他们需要新的基础设施——新的协作平台,新的代码框架,新的数据处理手段,新的算力使用方式;他们需要新的文化——追求通力协作、惠及大众;追求知识与工具的自由交流与分享;追求尊重并欣赏相互的成就、和而不同。”
——《DeepModeling社区宣言》

DeepModeling社区是这样的一群人的社区。DeepModeling Hackathon是这样的一群人的黑客马拉松!
赛题来啦!
赛题网址:
https://github.com/deepmodelinghackathon/hackathon2021
https://gitee.com/deepmodelinghackathon/hackathon2021
目前为赛题大纲预览,7月11日赛题最终确定,欢迎大家在此之前提出建议~
赛题总览:
我们提供了AI、科学计算、高性能计算三个赛道的相关题目,并且仍在持续更新中!同时飞桨(PaddlePaddle)团队也会带来相应的支持和指导(详情见赛题网址)。
AI方向
理解物理建模之智慧,洞若观火
1.探究原子邻域描述子的可解释性
2.抽取网络信息,实现模型压缩
3.神经网络架构搜索(NAS)
...
科学计算方向
打磨物理建模之利刃,吹毫立断
1.实现基于DP的热导、扩散系数等计算工作流设计
2.实现基于Abacus的材料能带计算工作流设计
3.FEALPy实现基于Bernstein多项式的有限元方法
...
高性能计算方向
将借物理建模之羽翼,直飞云空
1.Deepmd-kit混合精度训练优化
2.Abacus近邻原子搜索优化
3.FEALPy高效组装单元矩阵
...
Bonus题目(正式比赛放出,作为附加分数)
1.上手DeepMD-kit
2.上手PaddlePaddle
3.上手Abacus
...
赛制与评分规则(暂定):
即日起至正式比赛期间(8.15之前),我们会陆续放出部分赛题,比赛当天放出剩余题目。每队选手在报名截止(7.25)之前通过填写问卷的方式确定队伍的主赛道,并且选择1-2道题目作为赛前proposal(后续可更改),允许跨赛道选题。
每队于正式比赛当天确定1-2道题目,与最终proposal允许至多1道修改(但不允许更换主赛道),此部分题目的分数主要基于完成度和创新度;Bonus题目可选多道,不需要提前选定,可根据时间安排即答即交,我们根据此部分题目的完成度和题目数量非线性给分。
我们后续会通过线上直播的方式,向大家进行赛题介绍以及软件教学活动,帮助大家在赛前做好准备~
奖金在这里!!
每个赛道:
第一名 20000元
第二名 5000元
第三名 2000元
此外,所有参赛队伍都会获得定制专属纪念品!
活动安排
活动包括报告分享、项目教学、正式比赛与赛后颁奖观光。
免费报名,提供食宿,自理交通。
时间:8月16日—20日
地点:苏州
主办方:中科大苏州高等研究院
承办方:北京深势科技有限公司
日程:
赛前准备阶段(8月15日之前)
7月1日起:放出部分赛题,选手开始提交proposal及赛题意见
7月11日:更新最终赛题并确定数据集
7月12日起:开始线上直播软件教学(具体直播安排见后续推送)
7月25日:报名截止并停止提交proposal
(7月1日—8月15日是大家熟悉题目、开始动手研究题目的好时机哦)
活动开始(8月16日—20日,苏州)
8月16日—17日:报告分享与教学
聆听各位嘉宾的分享报告,学习使用相关软件工具,并随时交流答疑。
8月18日—20日:正式比赛
各组选定题目比赛,赛后专家团队评分,选出优秀队伍做展示并颁奖。
点击文末“阅读原文”获取活动介绍相关推送;讲座内容及具体评分标准见后续推送
Note:
1.可以只参与前两天的讲座与tutorial,后续三天的比赛为选择性报名。
2.比赛以自由组队或组委会帮助组队的形式,组成3-4人小队,也可以单人成队参赛;比赛中所用计算资源均由业内领先企业倾力提供。
3.比赛中推荐使用飞桨(PaddlePaddle)提供的深度学习插件,助力高效实现;如果使用PaddlePaddle,我们还会为你安排老师进行平台使用指导。
我要做什么?
1.填写报名问卷
复制此链接进行填写:
https://www.wjx.top/vm/toN7B3a.aspx
(之前填写过的同学不必重复填写,问卷以最新填写信息为准)。
2.提交赛题proposal及赛题反馈
复制此链接进行proposal提交与反馈:
https://www.wjx.top/vj/wbKckRf.aspx
(注:以上问卷均于7月25日23:59分截止)
欢迎扫码入群,了解详情
Q&A
Q:具体地点在哪里?
A:中科大苏州高等研究院。
Q:请问比赛可以线上吗?
A:为了比赛秩序以及及时答疑,原则上只允许现场参赛,若有特殊情况必须线上参赛,我们也会尽量满足大家。
Q:请问如何组队报名呢?是在报名表备注栏注明和XXX一组么?
A:选择参与比赛以后会出来组队选项,可以选择有合作队员,在后面写上队员名字就好。
Q:组队的话只需要一个人报名就行了?
A:最好是每个队员都填一份,把队友姓名写到合作队员里。
Q:请问报名听前两天讲座的可以当第三天的观众吗?
A:我们还是鼓励对比赛感兴趣的老师同学多多参与,也方便我们统一提供经济支持。
Q:网上报名分组填错了,还可以改吗?
A:可以重新填一下问卷,我们到时候会按最新的问卷结果来统计。
Q:报名人数过多会有参会资格筛选么?
A:我们尽量让大家都参加,如果人很多,也会提前和大家商量,最终的目的是促进大家交流、让大家都学到东西。
·DeepModeling社区开源协作平台·
http://github.com/deepmodeling
http://gitee.com/deepmodeling
·飞桨官网地址·
https://www.paddlepaddle.org.cn
关于飞桨
飞桨(PaddlePaddle)是中国首个开源最早、技术领先的产业级深度学习平台。此次比赛中,选手如果选择使用飞桨平台提供的深度学习插件,飞桨团队会为选手提供相应的技术支持以及平台使用指导,助力项目高效实现~
点击“阅读原文”跳转活动介绍。
飞桨(PaddlePaddle)以百度多年的深度学习技术研究和业务应用为基础,集深度学习核心训练和推理框架、基础模型库、端到端开发套件和丰富的工具组件于一体,是中国首个开源最早、技术领先的产业级深度学习平台。飞桨企业版针对企业级需求增强了相应特性,包含零门槛AI开发平台EasyDL和全功能AI开发平台BML。EasyDL主要面向中小企业,提供零门槛、预置丰富网络和模型、便捷高效的开发平台;BML是为大型企业提供的功能全面、可灵活定制和被深度集成的开发平台。
END

本文同步分享在 博客“飞桨PaddlePaddle”(CSDN)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。

AI+Science:基于飞桨的 AlphaFold2,带你入门蛋白质结构预测
1958 年 F.H.C. 克里克提出了生物学中重要的中心法则,DNA->RNA-> 蛋白质,中心法则说明,DNA 可以转录形成 RNA,RNA 再翻译成一个个氨基酸,最后组合形成蛋白质。
通过中心法则不难看出,如果把 DNA 比喻为进行工业生产的设计蓝图,那么蛋白质就像实现这个蓝图的工具,所以说蛋白质是一切生命活动的基础,它几乎参与了所有的生物学过程,如遗传、发育、繁殖等等。对蛋白质进行深入地研究,能让我们从更深层次诠释生命体的构成和运作变化规律,进而全面揭示生命运行、发展的机制,激发生物科学、药物研发、合成生物学、酶科学等领域的发展。
因探究生物体内各种蛋白质的功能及其机制等是目前蛋白质研究的主要内容,同时也是后基因组时代生命科学领域的主要研究热点之一。蛋白质的功能很大程度上取决于蛋白质的结构,因此如何破解蛋白质的三维结构成为了科学家研究的重点。
AlphaFold2 的诞生
近些年来,随着人工智能技术的发展,深度学习等相关技术也被应用在蛋白质结构预测领域。2018 年的 CASP 13(国际权威的蛋白质结构预测竞赛,每 2 年举办一次)上,谷歌 DeepMind 团队的 AlphaFold 拿下了 70 多分,打败众多研究团队,取得人工组第一,在该领域取得了里程碑式的进展。在 2020 年的 CASP 14 上,谷歌 DeepMind 团队的 AlphaFold2 以惊人的 92.4 分登顶第一 [1],这一结果也被认为是基本解决了 “困扰了生物学家 50 年” 的问题,获得重大突破。92.4 分,指的是对竞赛目标蛋白的预测精度 GDT_TS 分数达到 92.4,一般认为该分数超过 90 分,基本可以替代实验方式啦,这也意味着 AlphaFold2 预测的结果与实验得到的蛋白质结构基本一致。
2021 年 7 月 15 日, DeepMind 团队在国际顶级期刊《Nature》上发表论文,详细描述了 AlphaFold2 的设计思路,并提供了可供运行的基于 JAX 的模型和代码 [2]。考虑到 JAX 受众偏向专业的 AI 科学计算研究人员,且飞桨社区尚没有蛋白质结构预测相关的开源项目,百度螺旋桨 PaddleHelix 生物计算团队,基于飞桨深度学习框架,复现了 AlphaFold2 模型,提供给广大飞桨开发者使用,帮助大家快速入门蛋白质结构预测。
https://github.com/PaddlePaddle/PaddleHelix/tree/dev/apps/paddlefold
AlphaFold2 算法的
设计思路
AlphaFold2 通过独特的神经网络和训练过程设计,第一次端到端地学习蛋白质结构。整个算法框架通过协同学习蛋白质的多序列比对(MSA)和氨基酸对(pairwise)的表征,将蛋白质序列的进化信息、蛋白质结构的物理和几何约束信息结合到深度学习网络中。我们将从数据预处理、Evoformer 和 Structure Module 三个模块分析 AlphaFold2 算法的设计思想。
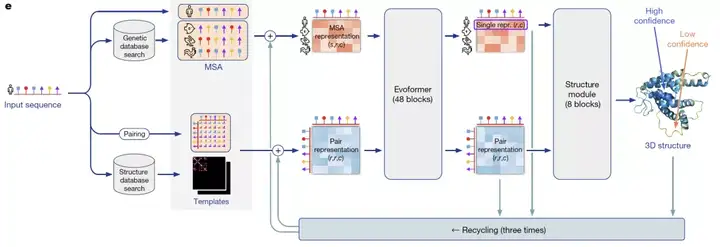
来自:AlphaFold2 论文
数据处理
预测蛋白结构时,AlphaFold2 会利用氨基酸序列信息在蛋白质库中搜索多序列比对(MSA)。MSA 可以反映氨基酸序列中的保守性区域(即不容易产生突变),这些保守性区域和蛋白质的结构息息相关,比如可能被折叠在蛋白质内层,不容易和外界产生相互作用,进而不易受影响发生突变。在 AlphaFold2 的数据预处理中,为了减少模型运算量,会先对 MSA 中的序列进行聚类,取每个类别中心的序列作为 main MSA 特征。除了 MSA,AlphaFold2 的另一个重要输入是氨基酸对(pairwise)的特征。作为 main MSA 的补充,Alphafold2 会随机采样非聚类中心的序列作为 extra MSA 输入一个 4 层的网络提取 pairwise 特征,然后和模版提取的 pairwise 特征相加后得到最终 pairwise 特征。main MSA 特征和 pairwise 特征通过 48 层 Evoformer 进行表征融合。
Evoformer
Evoformer 网络的设计动机是想利用 Self-Attention 机制学习蛋白质的三角几何约束信息,同时让 MSA 表征带来的共进化信息和 pairwise 表征的结构约束信息相互影响,使得模型能直接推理出空间信息和进化信息的联系。
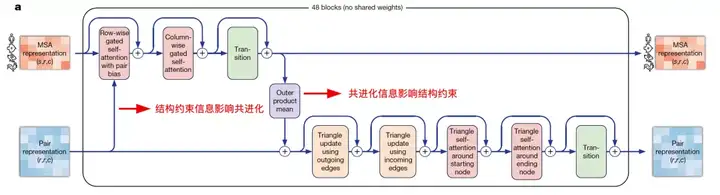
来自:AlphaFold2 论文
Structure Module
Structure Module 承担着把 Evoformer 得到的表征解码成蛋白质中每个重原子 (C,N,O,S) 坐标的任务。为了简化从神经网络预测值到原子坐标的转换,AlphaFold2 结合蛋白质中 20 类氨基酸的结构特性,将重原子分成不同二面角转角决定的组,这样就可以根据给定的起始位置,利用二面角和氨基酸已知的键长键角信息解码出原子坐标。这种结构编码方法相比直接预测坐标 (x,y,z) 大大降低了神经网络的预测空间,使得端到端结构学习成为可能。

赖氨酸的转角编码方式示例:蓝色平面(C,Cα,Cβ)确定后,根据预测的蓝色 - 紫色平面的二面角 χ1 和已知的 C-C 键长,Cγ-Cβ-N 键角即可确定 Cγ 的空间坐标,重复类似步骤,可以得到 Cδ,Cε, N 等重原子坐标。
基于飞桨框架的
AlphaFold2(AF2)使用
目前已经基于飞桨框架复现了完整的 AlphaFold2 的 inference 部分,现已正式在螺旋桨 PaddleHelix 平台开源:https://github.com/PaddlePaddle/PaddleHelix/tree/dev/apps/paddlefold
感兴趣的小伙伴们可以安装使用,并基于此,优化自己的蛋白结构预测模型。
1. 安装
在 requirements.txt 中提供了通过 pip 可安装的 Python 依赖项。另外,(基于飞桨框架的 AF2 还依赖于两个只能通过 conda 安装的 工具包:openmm==7.5.1 和 pdbfixer。为了得到多序列比对 MSA,还需要安装 kalign, HH-suite 和 jackhmmer。下载数据的脚本需要 aria2c。
提供一个可以设置 conda 环境并安装所有依赖项的脚本 setup_env。运行:
sh setup_env
conda activate paddlefold # activate the conda environment
也可以在 setup_env 中更改环境名称和 CUDA 版本。
2. 用法
为了运行基于飞桨框架的 AF2,还需要蛋白序列数据库和模型参数。基于飞桨框架的 AF2 使用和 AlphaFold2 一样的模型参数。
你可以使用脚本 scripts/download_all_data.sh 来下载和设置所有数据库和模型参数。
- 运行:
scripts/download_all_data.sh <DOWNLOAD_DIR>
将下载完整的数据库。完整数据库的总下载大小约为 415 GB,解压后的总大小为 2.2 TB。
- 运行:
scripts/download_all_data.sh <DOWNLOAD_DIR> reduced_dbs
将下载一个减少版本的数据库,可以用于在 reduced_ dbs 的设置下运行。减少的数据库的总下载大小约为 190GB,解压缩后的总下载大小约为 530GB。
3. 运行基于飞桨框架的 AF2 进行推理
要使用 DeepMind 已经训练好的参数对一个序列或多个序列进行推理,运行例如:
fasta_file="target.fasta" # path to the target protein
model_name="model_1" # the alphafold model name
DATA_DIR="data" # path to the databases
OUTPUT_DIR="paddlefold_output" # path to save the outputs
python3 run_paddlefold.py \
--fasta_paths=${fasta_file} \
--data_dir=${DATA_DIR} \
--small_bfd_database_path=${DATA_DIR}/small_bfd/bfd-first_non_consensus_sequences.fasta \
--uniref90_database_path=${DATA_DIR}/uniref90/uniref90.fasta \
--mgnify_database_path=${DATA_DIR}/mgnify/mgy_clusters_2018_12.fa \
--pdb70_database_path=${DATA_DIR}/pdb70/pdb70 \
--template_mmcif_dir=${DATA_DIR}/pdb_mmcif/mmcif_files \
--obsolete_pdbs_path=${DATA_DIR}/pdb_mmcif/obsolete.dat \
--max_template_date=2020-05-14 \
--model_names=${model_name} \
--output_dir=${OUTPUT_DIR} \
--preset=''reduced_dbs'' \
--jackhmmer_binary_path /opt/conda/envs/paddlefold/bin/jackhmmer \
--hhblits_binary_path /opt/conda/envs/paddlefold/bin/hhblits \
--hhsearch_binary_path /opt/conda/envs/paddlefold/bin/hhsearch \
--kalign_binary_path /opt/conda/envs/paddlefold/bin/kalign \
--random_seed=0
你可以使用 python3 run_paddlefold.py -h 来查找参数的描述。
保留与 AlphaFold2 相同的输出,输出将位于 output_dir 的子文件夹中。它们包括计算的 MSAs、模型预测的蛋白结构、OpenMM 优化后的结构、模型打分排序、原始模型输出、预测元数据和模型运行计时。output_dir 目录将具有以下结构:
<target_name>/
features.pkl
ranked_{0,1,2,3,4}.pdb
ranking_debug.json
relaxed_model_{1,2,3,4,5}.pdb
result_model_{1,2,3,4,5}.pkl
timings.json
unrelaxed_model_{1,2,3,4,5}.pdb
msas/
bfd_uniclust_hits.a3m
mgnify_hits.sto
uniref90_hits.sto
每个输出文件的内容如下:
- features.pkl
一个 pickle 文件,其中包含模型用于生成结构的输入特性 NumPy 数组。 - unrelaxed_model_*.pdb
一个 PDB 格式的文本文件,其中包含预测的结构,与模型输出的结构完全一样。 - relaxed_model_*.pdb
一个 PDB 格式的文本文件,是调用 OpenMM 得到的优化结构,修复了模型预测结构中的冲突,并添加 H 原子的坐标位置。 - ranked_*.pdb
一个 PDB 格式的文本文件,是对 OpenMM 得到的优化结构按照模型置信度的重新排序。这里使用预测的 LDDT 分数 (pLDDT) 作为置信度评估。 - ranking_debug.json
一个 JSON 格式的文本文件,包含用于执行模型排名的 pLDDT 值及其对应的模型名称。 - timings.json
一个 JSON 格式的文本文件,包含运行 AlphaFold2 模型的每个部分所花费的时间。 - msas/
该目录中包含不同 MSA 搜索工具的输出文件。 - result_model_*.pkl 一个 pickle 文件,其中包含一个由模型直接生成的各种 NumPy 数组的字典,除了结构模块的输出外,还包括辅助输出。
最后,可以使用 pymol [3] 等工具对预测结构和实验结构对齐。值得说明的是,由于输入特征存在采样操作,基于飞桨框架复现的 AlphaFold2 和 JAX 版本的预测结构可能会有略微差异,有时候会和实验结构更接近,也可能差别稍大。
近期开发计划
AlphaFold2 虽然在单体蛋白上表现优异,但对复合体,预测的准确度还有待提升。为此,DeepMind 团队上线了 AlphaFold-Multimer 模型,一款针对复合物进行重新训练的神经网络模型,希望能发动飞桨社区开发者们的积极性,一起开发优化基于 AlphaFold-Multimer 的模型,之后也开源贡献到飞桨平台,让更广大的生信领域研究者们使用基于飞桨框架完全自主可控的蛋白结构预测模型。
参考文献 [1] https://predictioncenter.org/casp14/zscores_final.cgi.[2]Jumper, J.; Evans, R.; Pritzel, A.; Green, T.; Figurnov, M.; Ronneberger, O.; Tunyasuvunakool, K.; Bates, R.; Zidek, A.; Potapenko, A.; Bridgland, A.; Meyer, C.; Kohl, S. A. A.; Ballard, A. J.; Cowie, A.; Romera-Paredes, B.; Nikolov, S.; Jain, R.; Adler, J.; Back, T.; Petersen, S.; Reiman, D.; Clancy, E.; Zielinski, M.; Steinegger, M.; Pacholska, M.; Berghammer, T.; Bodenstein, S.; Silver, D.; Vinyals, O.; Senior, A. W.; Kavukcuoglu, K.; Kohli, P.; Hassabis, D., Highly accurate protein structure prediction with AlphaFold. Nature 2021, 596, 583-589.[3] https://pymol.org
关于CSC1001: Introduction to Computer Science的介绍现已完结,谢谢您的耐心阅读,如果想了解更多关于AI for Science 交流会来了!科学计算前沿邀您共同探讨、AI+Science 系列(二):国内首个基于 AI 框架的 CFD 工具组件!赛桨 v1.0 Beta API 介绍以及典型案例分享!、AI+Science 黑客马拉松|赛程发布|10万奖金等你来拿!、AI+Science:基于飞桨的 AlphaFold2,带你入门蛋白质结构预测的相关知识,请在本站寻找。
本文标签: